Avances en la Simulación de Proteínas mediante Aprendizaje Automático
Un equipo internacional liderado por la profesora Cecilia Clementi, del Departamento de Física de la Freie Universität Berlin, ha realizado un descubrimiento significativo en el campo de la simulación de proteínas. Este estudio, publicado en la revista Nature Chemistry, introduce el modelo CGSchNet, un sistema basado en aprendizaje automático que promete revolucionar la manera en que se simulan las proteínas.
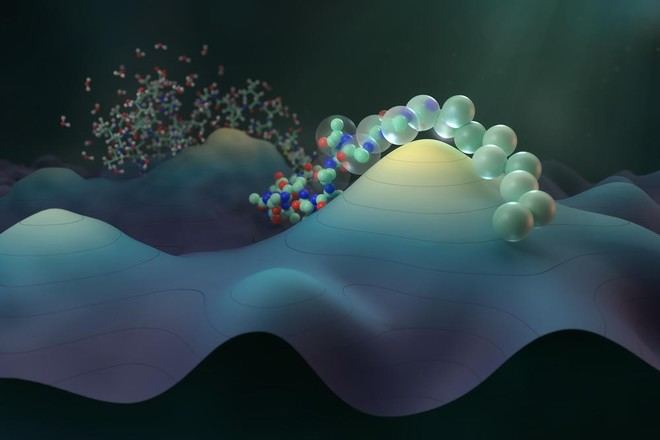
CGSchNet es un modelo de grano grueso (CG) que puede simular proteínas con una precisión y eficiencia sin precedentes. A diferencia de los métodos tradicionales de dinámica molecular que operan a nivel atómico, CGSchNet funciona a una velocidad considerablemente mayor, permitiendo así explorar proteínas más grandes y sistemas complejos. Esto abre nuevas posibilidades para aplicaciones en el descubrimiento de fármacos y la ingeniería de proteínas, lo cual podría tener implicaciones importantes en el tratamiento del cáncer.
Un Avance Clave en la Simulación de Dinámicas Proteicas
Durante más de cincuenta años, desarrollar un modelo general capaz de capturar el plegamiento y las dinámicas proteicas ha sido un desafío constante para los científicos. Según Cecilia Clementi, “este trabajo es el primero en demostrar que el aprendizaje profundo puede superar esta barrera y llevar a un sistema de simulación que aproxima las simulaciones atómicas sin modelar explícitamente el solvente o los detalles atómicos”.
El equipo entrenó una red neuronal gráfica para aprender las interacciones efectivas entre las partículas del modelo CG, logrando reproducir la dinámica de miles de simulaciones atómicas. A diferencia de las herramientas convencionales de predicción estructural, CGSchNet modela procesos dinámicos, incluyendo estados intermedios relevantes para fenómenos como el mal plegamiento, asociado a agregados patológicos como los amiloides presentes en enfermedades como Alzheimer.
Además, este innovador modelo no solo simula transiciones entre estados plegados —esenciales para la función proteica— sino que también generaliza a proteínas fuera del conjunto utilizado para su entrenamiento, mostrando una notable transferencia química. Su capacidad para predecir estados metastables y energías libres relativas del plegamiento representa un avance significativo frente a limitaciones computacionales anteriores.
Cecilia Clementi: Pionera en Biología Computacional
La profesora Cecilia Clementi es una destacada biofísica teórica y computacional. Antes de su llegada a Berlín en 2020, fue profesora de química y física en Rice University, Texas. Su posición en Freie Universität refuerza la investigación en biología física teórica y asistida por computadora en Berlín, creando un puente entre la biología experimental y las matemáticas aplicadas.
Este progreso no solo destaca por sus implicaciones científicas sino también por el apoyo institucional recibido a través del programa de Becas Einstein, diseñado para atraer y retener científicos pioneros en Berlín.

 Si (
Si ( No(
No(











